Kapitel 5 Daten einlesen und visualisieren
- Daten aus Textdateien in R einlesen
- Grafiken anpassen (nebeneinander, Facetten, Transparenz)
- Grafiken speichern
5.1 Daten aus Textdateien in R einlesen
Um Daten aus Textdateien (z.B. aus .csv, .txt, .dat) in R zu importieren (i.e. einzulesen) werden wir die Bibliothek readr aus tidyverse benutzen. Wir laden erst einmal tidyverse.
Wir gehen davon aus, dass die Daten im Ordner data gespeichert sind. Falls Ihre Daten an einem anderen Ort abgelegt sind, müssen Sie den Pfad beim Einlesen entsprechend anpassen.
Um die Daten zu laden, gibt es in der Bibliothek readr verschiedene Funktionen, die alle mit read_ beginnen. Die allgemeinste davon ist read_delim. Darin kann man explizit einstellen, mit welchem Zeichen (z.B. Komma, Strichpunkt etc.) die einzlenen Spalten in der zu importierenden Datei getrennt sind.
emissions <- read_delim(file = 'data/emissions.csv', delim = ';')## Rows: 2871 Columns: 6
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ";"
## chr (4): unit, airpol, vehicle, geo
## dbl (1): values
## date (1): time
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Ein kurzer Blick auf den Datensatz. Hierbei handelt es sich um Daten zu Treibhausgasemissionen auf der EU-Ebene, die ich bei eurostat am 30.4.2021 heruntergeladen und vorgefiltert habe. Die Datenbank bietet sehr viele Datensäzte und ist als Quelle für Berichte hervorragend geeignet 😄.
emissions## # A tibble: 2,871 × 6
## unit airpol vehicle geo time values
## <chr> <chr> <chr> <chr> <date> <dbl>
## 1 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Aust… 2018-01-01 14.4
## 2 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Belg… 2018-01-01 14.4
## 3 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Bulg… 2018-01-01 5.78
## 4 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Swit… 2018-01-01 11.0
## 5 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Cypr… 2018-01-01 1.38
## 6 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Czec… 2018-01-01 11.9
## 7 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Germ… 2018-01-01 97.8
## 8 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Denm… 2018-01-01 6.85
## 9 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Esto… 2018-01-01 1.52
## 10 Million tonnes Greenhouse gases (CO2, N2O in… Fuel c… Gree… 2018-01-01 7.61
## # … with 2,861 more rowsDas Ergebnis des Einlesens mit read_ Funktionen ist immer ein tibble. Kategorische Variablen werden als Text (character) eingelesen und nicht in factor umgewandelt. Wenn man factor möchte, muss man die Variablen per Hand umwandeln.
Wir verschaffen uns einen kurzen Überblick über die Daten.
summary(emissions)## unit airpol vehicle geo
## Length:2871 Length:2871 Length:2871 Length:2871
## Class :character Class :character Class :character Class :character
## Mode :character Mode :character Mode :character Mode :character
##
##
##
##
## time values
## Min. :1990-01-01 Min. : 0.00609
## 1st Qu.:1997-01-01 1st Qu.: 0.25564
## Median :2004-01-01 Median : 1.92403
## Mean :2004-01-01 Mean : 8.52836
## 3rd Qu.:2011-01-01 3rd Qu.: 6.93899
## Max. :2018-01-01 Max. :119.77824
## NA's :232Um die Anzahl der einzelnen Länder zu ermitteln, sehen wir uns die Länge der Ausgabe der Funktion unique() an, die die einzelnen verschiedenen Einträge ermitteln kann. Es sind Einträge für 33 verschiedene Länder vorhanden.
## [1] 335.2 Legende verschieben und Facetten
Wir stellen die Zeitreihen der Emissionen eingefärbt nach Land dar. Die Länder stehen in der Variablen geo.
## Warning: Removed 7 row(s) containing missing values (geom_path).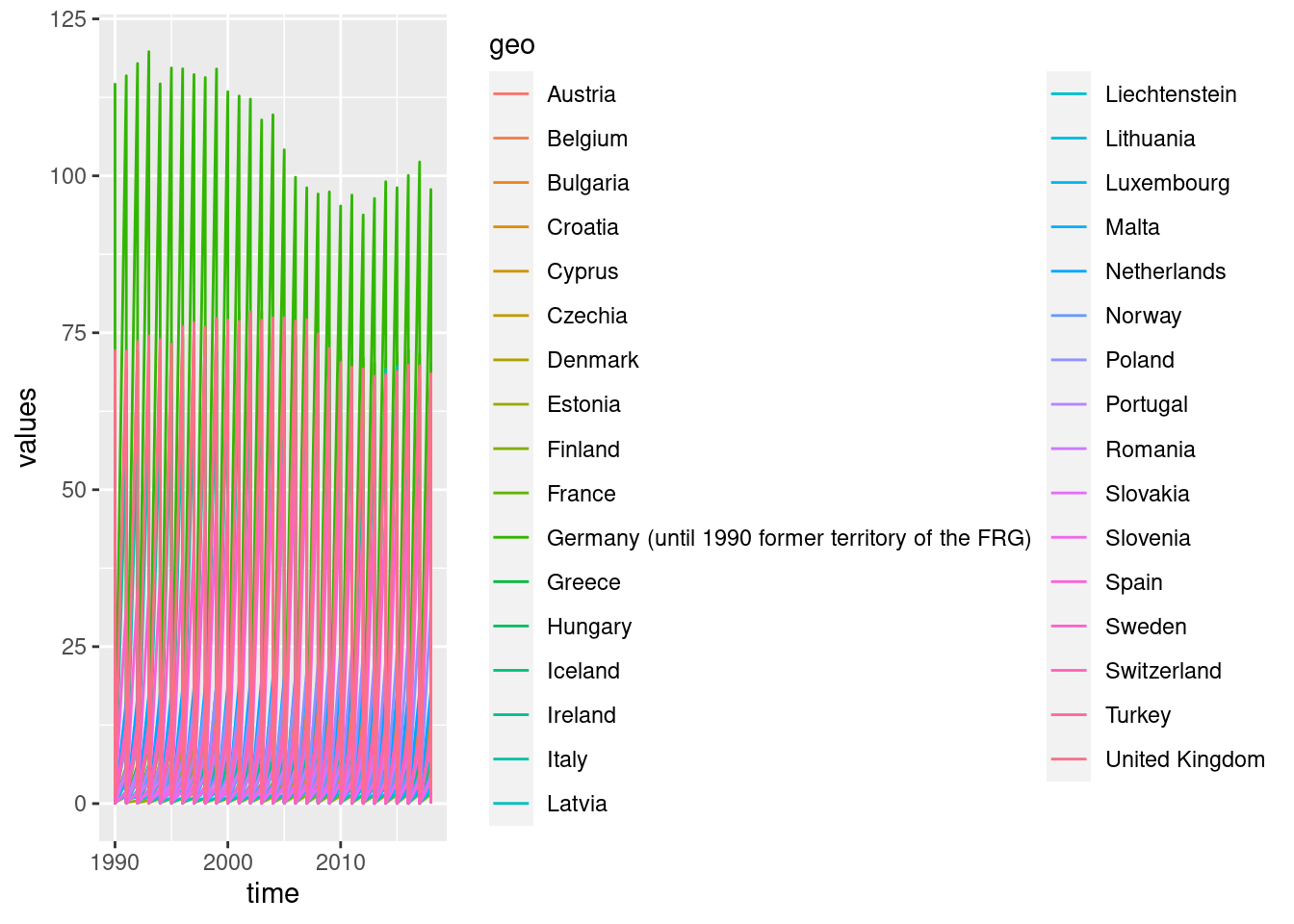
Als erstes fällt auf, dass die Legende sehr umfangreich ist (wir haben ja 33 Länder im Datensatz). Daher wäre es günstig, die Legende unterhalb der Grafik zu positionieren und den Titel der Legende oberhalb der Legende zu belassen. Das geht mit Hilfe der Funktionen theme() und guides(). Wie immer, werden sie im Plotaufbau (denken Sie grammer of graphics) mit + angehängt.
ggplot(data = emissions, mapping = aes(x = time, y = values, colour = geo)) +
geom_line() +
theme(legend.position = "bottom") +
guides(colour = guide_legend(title.position = "top"))## Warning: Removed 7 row(s) containing missing values (geom_path).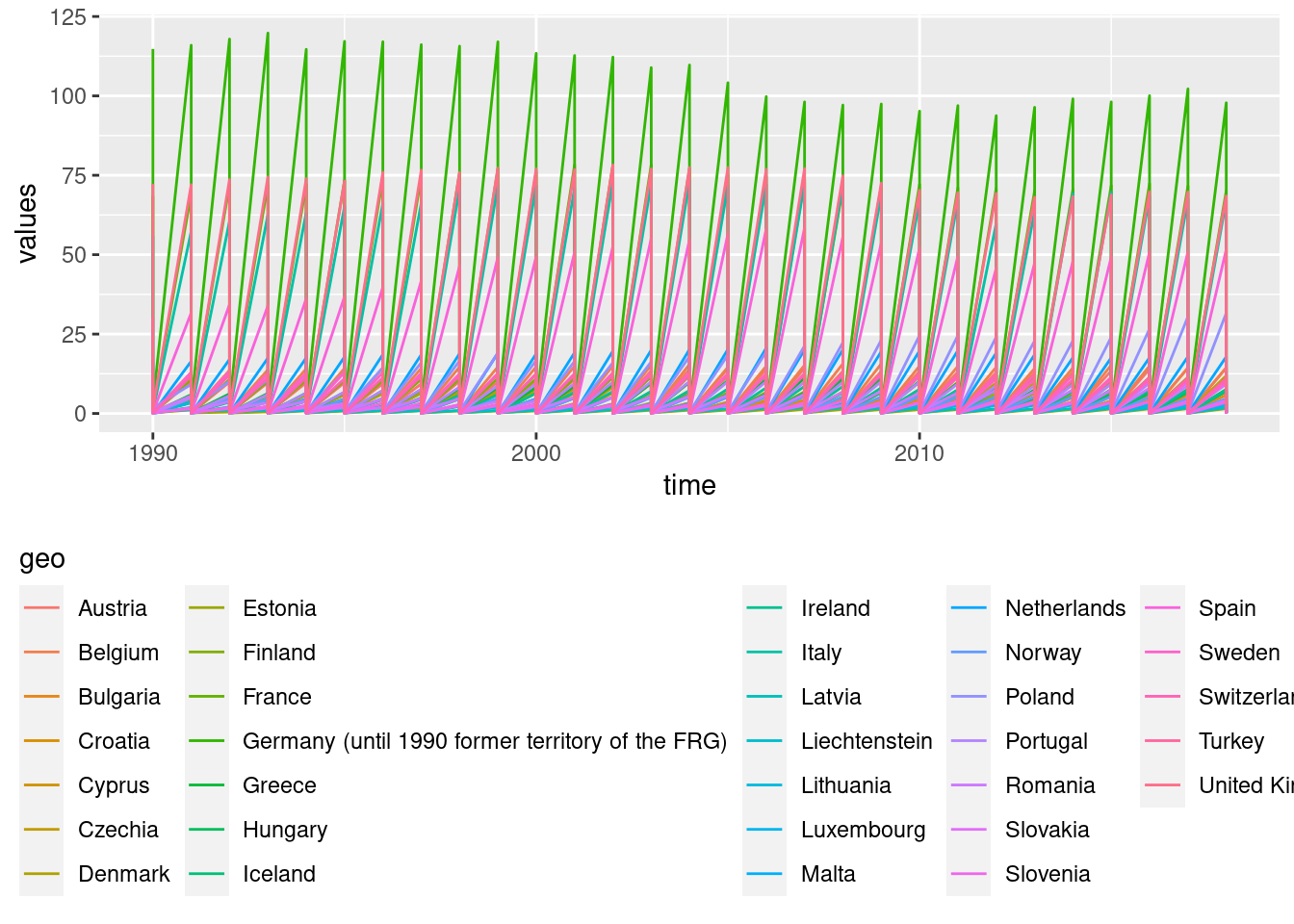
Die Zeitreihen sehen echt seltsam aus. Wenn wir uns die Variable vehicle ansehen, wird auch klar, warum. Wir stellen gerade Emissionen für verschiedene Fahrzeuge dar, d.h. wir mischen mehrere Zeitreihen zusammen.
unique(emissions$vehicle)## [1] "Fuel combustion in cars"
## [2] "Fuel combustion in heavy duty trucks and buses"
## [3] "Fuel combustion in railways"Die einfachste Lösung ist, drei verschiedene Grafiken pro Verkehrsmittel zu erstellen. Dies gelingt sehr leicht mit der Funktion facet_wrap(), die den Namen der Variablen erwartet, mit Hilfe derer die Grafiken gesplittet werden sollen. Vor der Variablen muss eine Tilde (~) stehen. In unserem Fall wollen wir nach Verkehrsmittel splitten, d.h. mit Hilfe der Varialben vehicle.
ggplot(data = emissions, mapping = aes(x = time, y = values, colour = geo)) +
geom_line() +
facet_wrap(~vehicle) +
theme(legend.position = "bottom") +
guides(colour = guide_legend(title.position = "top"))## Warning: Removed 203 row(s) containing missing values (geom_path).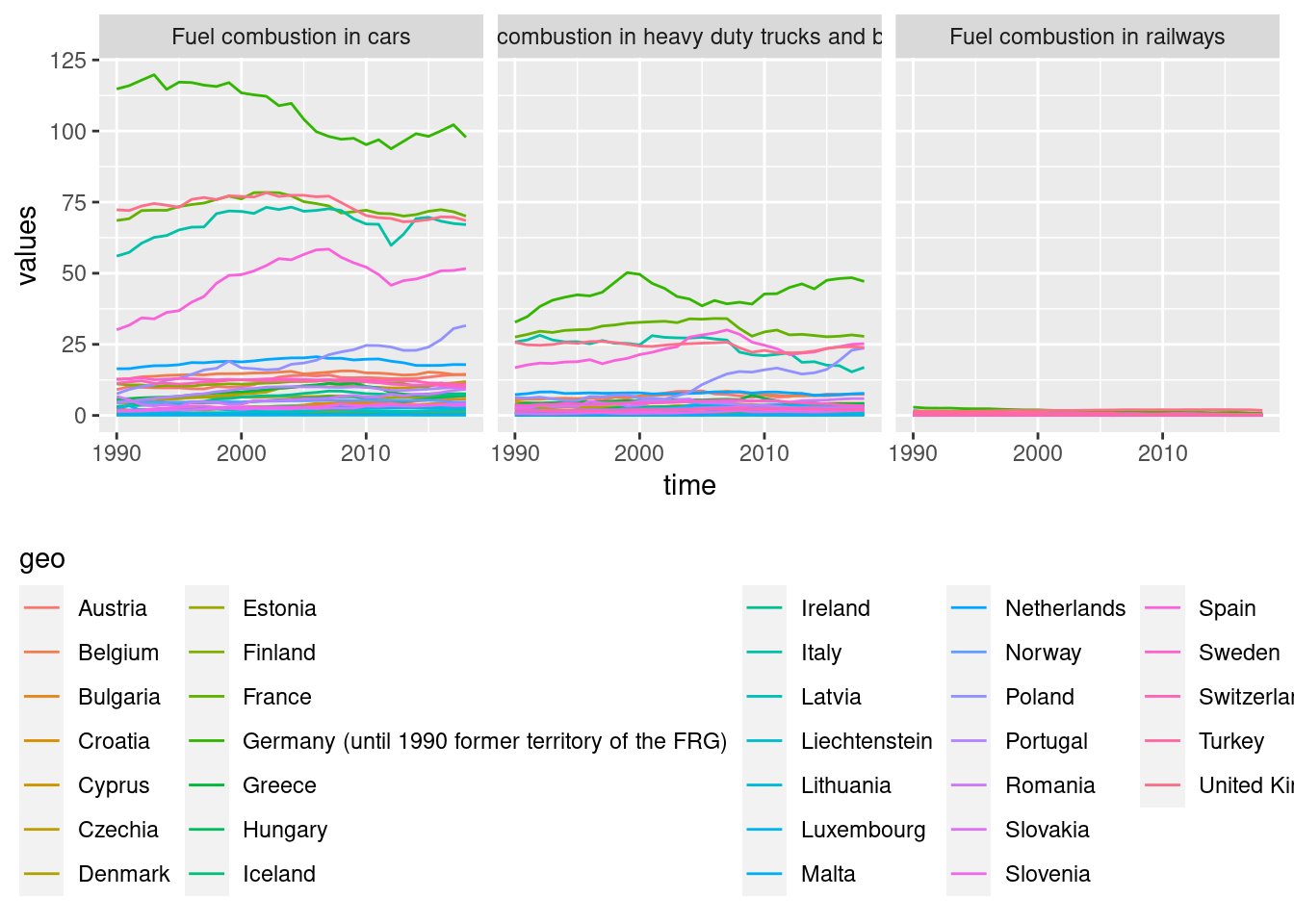
Da die Emissionen sehr unterschiedlich sind, macht es Sinn, die Skalierungen der y-Achsen anzupassen. Aber Achtung: Das sollten Sie in Ihren Berichten unbedingt ansprechen (z.B. in der Bildunterschrift), da man unterschiedliche Skalierunge sehr leicht übersieht und dann die Interpretation der Daten leicht in die falsche Richtung gehen kann. Der Funktionsparameter labeller = label_wrap_gen() sorgt für geschickte Zeilenumbrüche bei zu langen Labels. Zum Vergleich können Sie ihn mal weglassen und sehen, was dann passiert.
ggplot(data = emissions, mapping = aes(x = time, y = values, colour = geo)) +
geom_line() +
facet_wrap(~vehicle, scales = 'free_y', labeller = label_wrap_gen()) +
theme(legend.position = "bottom") +
guides(colour = guide_legend(title.position = "top"))## Warning: Removed 203 row(s) containing missing values (geom_path).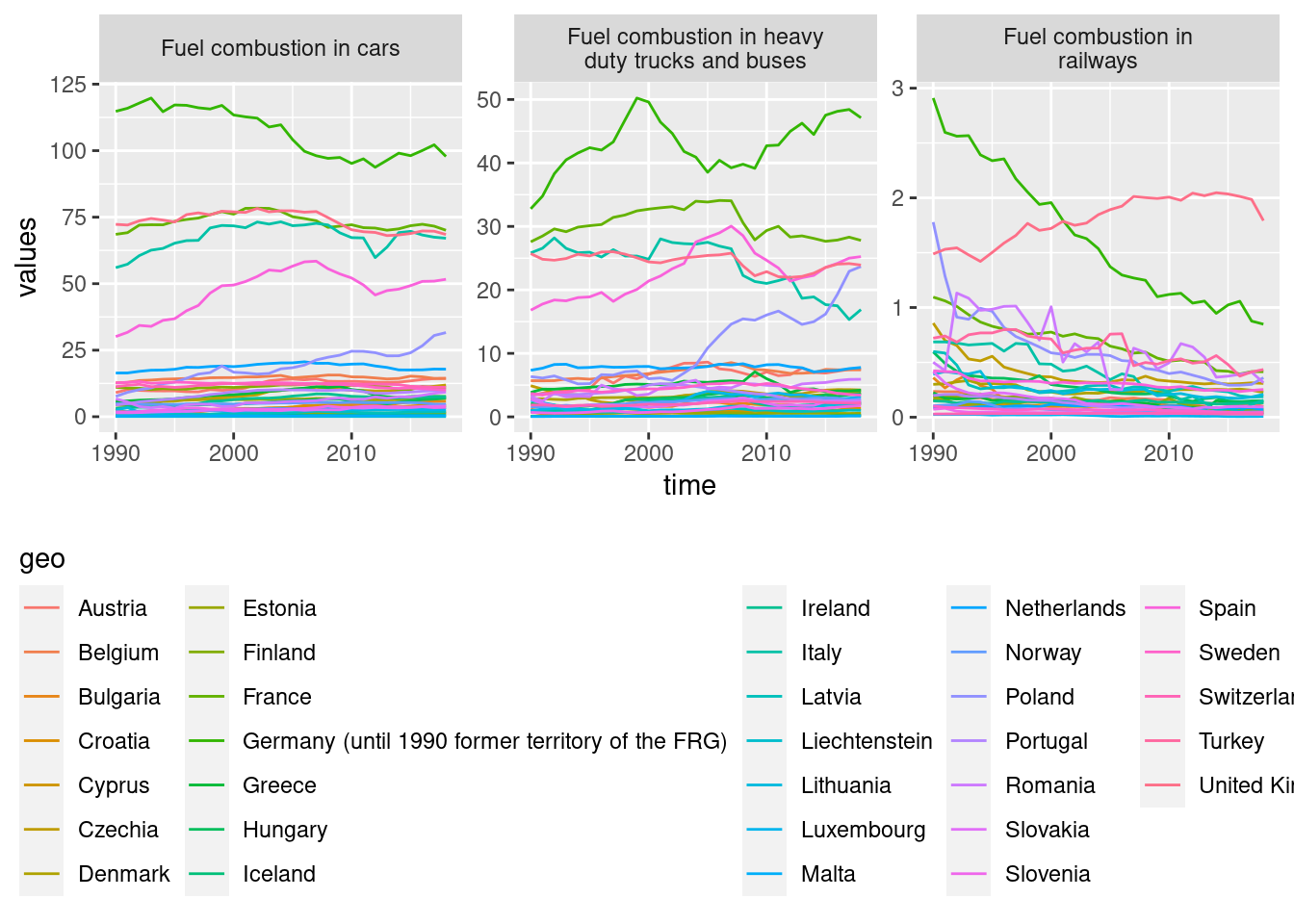
5.3 Fehlerbalken und Co.
Um die Variabilität der Daten grafisch darzustellen, bieten sich Fehlerbalken, Bereiche etc. an. Dafür hat ggplot2 spezielle geoms. Hier ein Beispiel inspiriert vom dem Buch ggplot2 (Wickham 2020).
y <- c(10, 5, 23)
df <- tibble(x = 1:3, y = y, se = c(0.9, 1.5, 3.3))
basis_plot <- ggplot(df, aes(x, y, ymin = y - se, ymax = y + se))
basis_plot + geom_pointrange()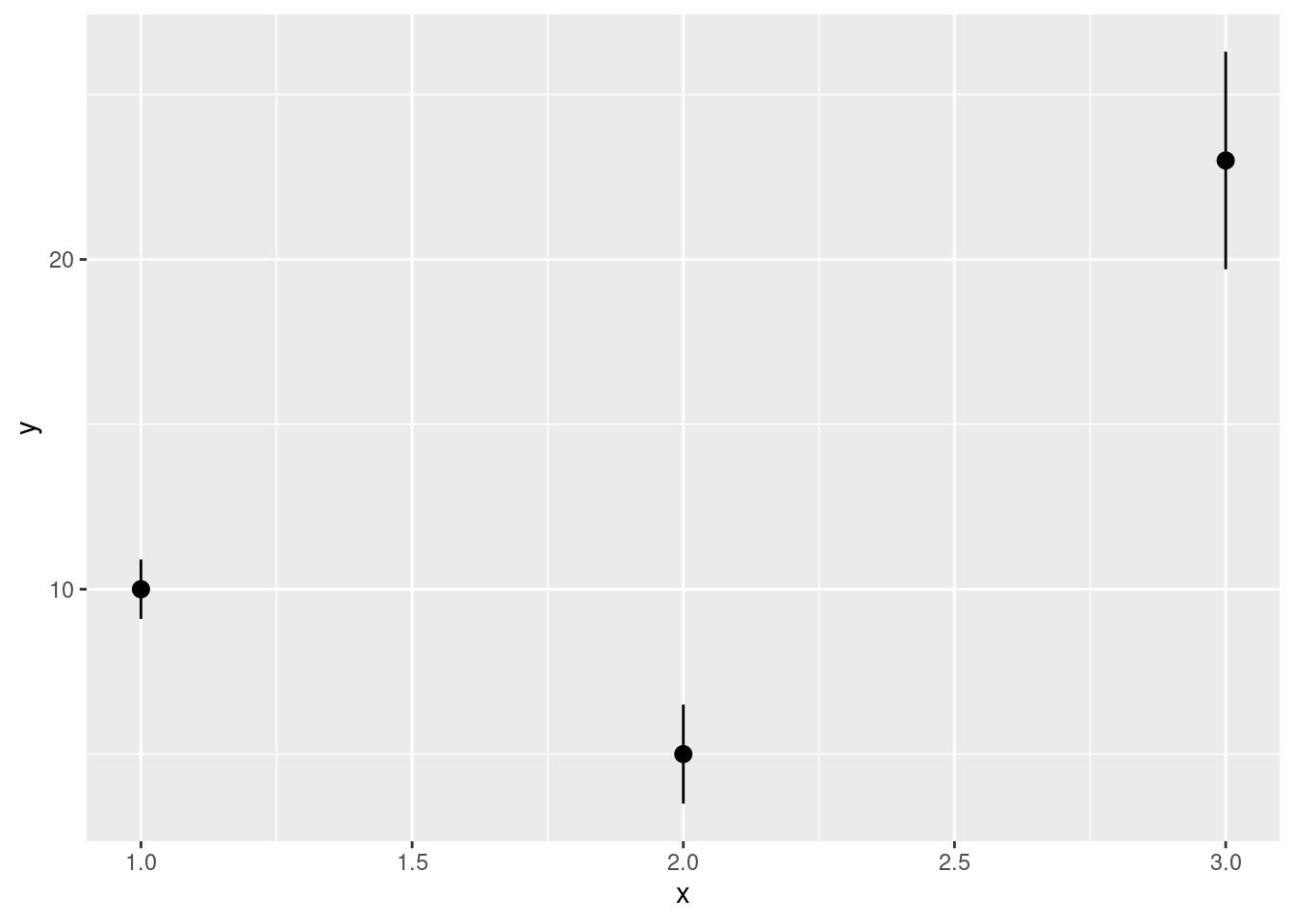
basis_plot + geom_errorbar()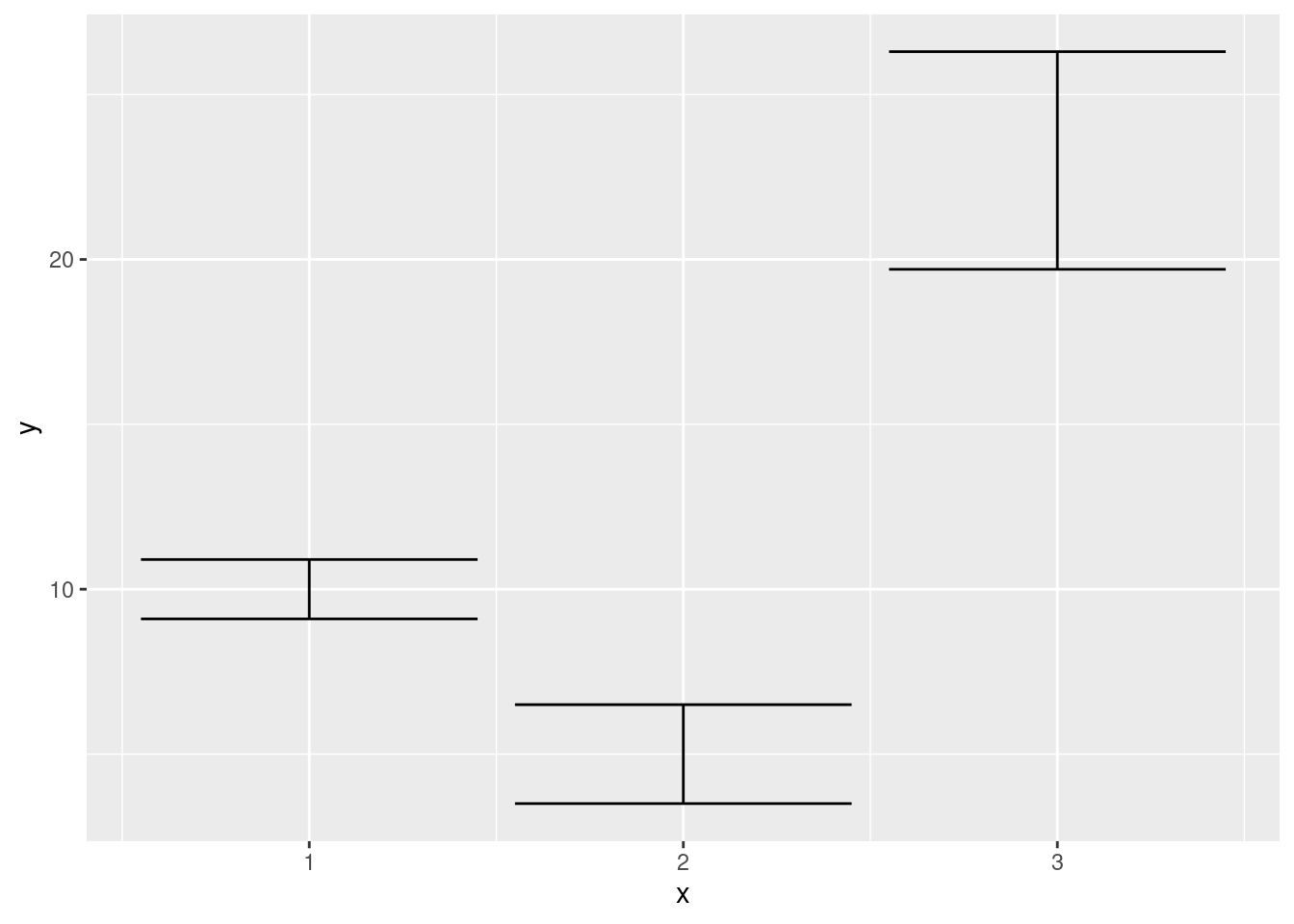
Sie sehen, dass man ggplot Objekte so wie andere Objekte in R zuweisen kann, um mit ihnen später zu arbeiten. In diesem Fall ist basis_plot ein ggplot Objekt.
class(basis_plot)## [1] "gg" "ggplot"5.4 Mehrere Grafiken nebeneinander
Um mehrere Grafiken nebeneinander zu plotten, nutzen wir die Bibliothek patchwork. Sie erlaubt verschieden Layouts. Details finden Sie hier. Die einfachsten Varianten sind zwei Grafiken nebeneinander. Dafür erstellen wir zwei ggplot-Objekte und verbinden sie mit einem +.
library(patchwork)
p1 <- basis_plot + geom_pointrange()
p2 <- basis_plot + geom_errorbar()
p1 + p2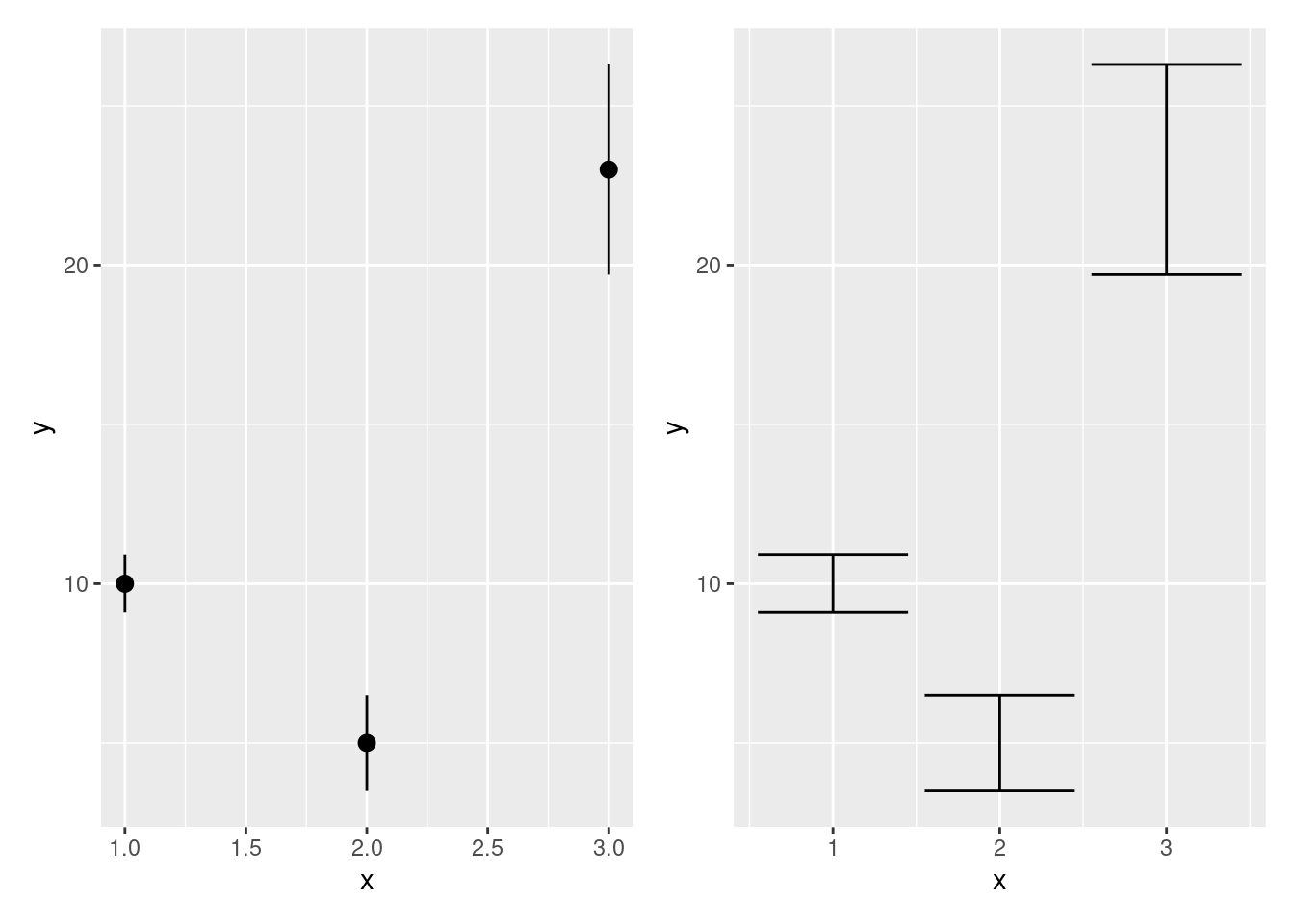
Um die Grafiken untereinander abzubilden, nutzen wir das Zeichen /.
p1 / p2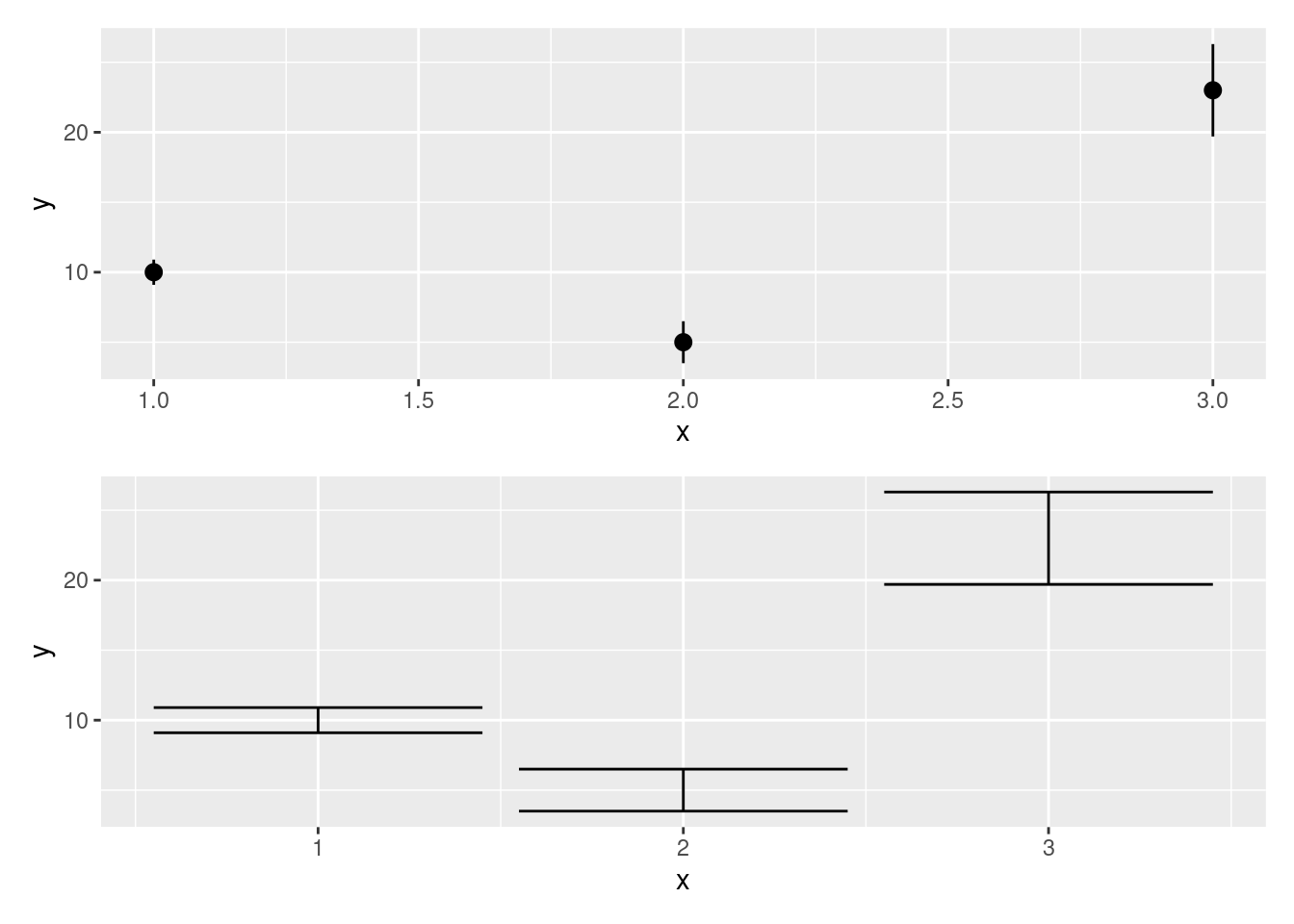
5.5 Grafiken abspeichern
Die Funktion ggsave() speichert ggplot Grafiken ab. Wenn nicht anders angegeben wird, wird der letzte aktuelle Plot gespeichert.
ggsave(filename = 'Fehlerbalken.pdf', device = 'pdf', width = 7, height = 5)Wir können aber auch explizit ein ggplot Objekt zum Speichern benennen.
alles <- p1 + p2
ggsave(filename = 'Fehlerbalken_2.pdf', device = 'pdf', plot = alles, width = 7, height = 5)5.6 Weitere statistsiche Zusammenfassungen in Grafiken
Arbeiten Sie selbständig das Kapitel 5: Statistical summaries in Wickham (2020) (https://ggplot2-book.org/statistical-summaries.html).
5.7 Lesestoff
Kapitel 2.2 bis 2.9 in Ismay and Kim (2021)
5.8 Aufgaben
5.8.1 Grafiken richtig beschriften
Beschriften Sie die finale Grafik der Zeitreihen (Achsen, Titel, Legende).
5.8.2 Bestandesaufnahme im Wald
Ar Stat arbeitet als HiWi in der AG Ökosystemforschung und soll im Nationalpark Eifel eine Bestandsaufnahme durchführen (d.h. Baumhöhen und -durchmesser vermessen). Er notiert den BHD (Brusthöhendurchmesser) und die Art der Bäume.
- Lesen Sie den Datensatz BHD.txt ein und ordnen Sie ihn der Variable
BHDzu. - Erstellen Sie einen Vektor
Nrmit durchlaufenden Baumnummern. Von welcher Art sind die Elemente des VektorsNr? Tipp: Verwenden Sie das Zeichen:, um durchlaufende Nummern zu erzeugen. - Fügen Sie die Datensätze
BHDundNrzu einemtibblezusammen und benennen Sie die Spalten dabei sinnvoll. - Löschen Sie den Vektor
Nr. Tipp: Hilfe zu Funktionrm()lesen. - Lesen Sie den Datensatz Art.txt ein und ordnen Sie ihn der Variablen
artzu. - Fügen Sie die Art in das
tibbleein. Tipp:tibble(altes_tibble, neue_spalte = XY). - Erstellen Sie eine Tabelle mit der Anzahl der jeweiligen Arten. Nutzen Sie die Funktion
table(). - Speichern Sie die Tabelle mit
write_delim()ab. Schlagen Sie in der Hilfe nach, wie diese Funktion arbeitet! Tipp: Vorher muss der Datensatz zu einem data.frame umgewandelt werden. Nutzen Sie die funktionas.data.frame().
5.8.3 Wahlbeteiligung bei der Bundestagswahl 2017
Bauen Sie die Grafik nach (Abbildung 5.1).
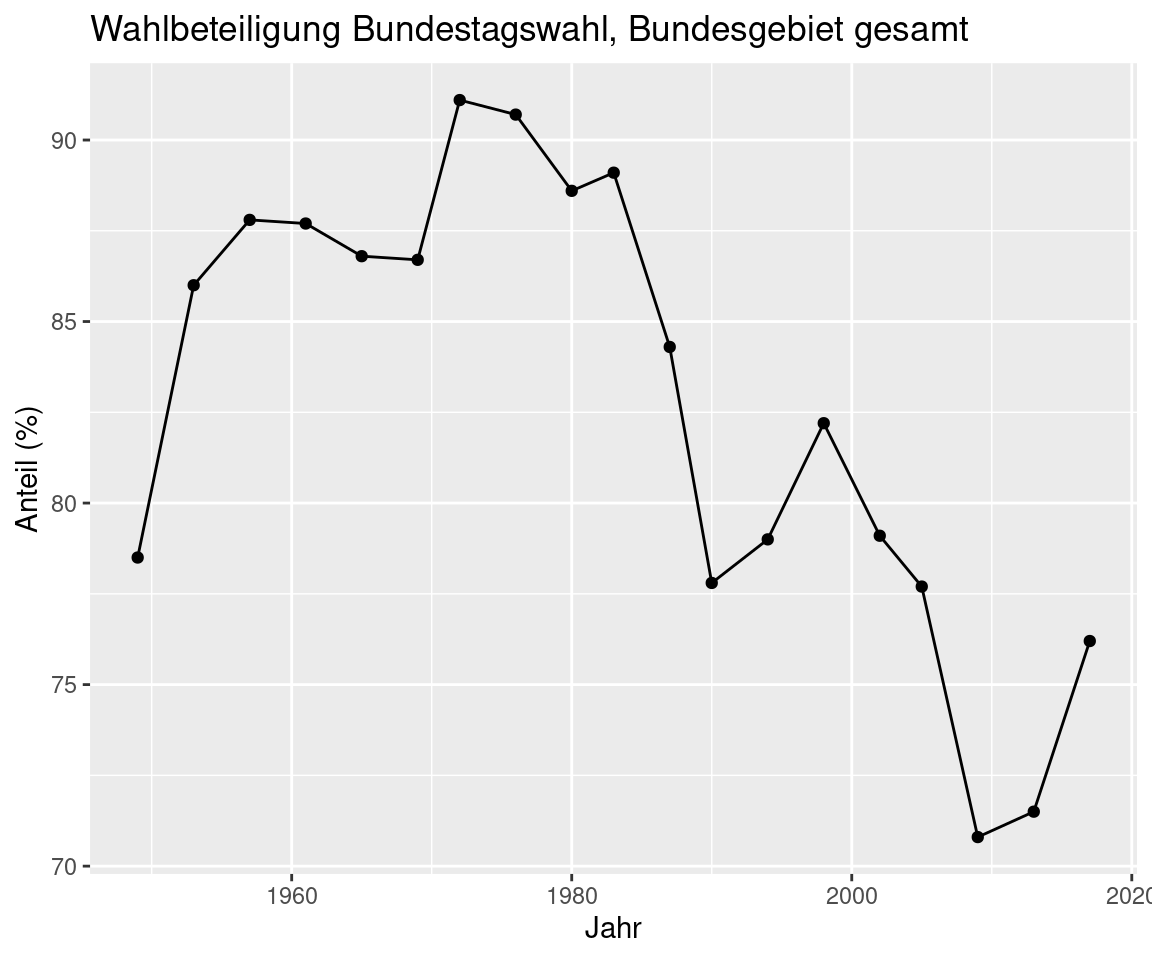
Abbildung 5.1: Wahlbeteiligung bei den Bundestagswahlen. Quelle: Der Bundeswahlleiter.
- Lesen Sie den Datensatz Wahlbeteiligung.csv in R ein und ordnen Sie ihn dem Objekt
beteiligungzu. - Sehen Sie sich den Datensatz an und fassen Sie ihn zusammen.
- Stellen Sie die Wahlbeteiligung als Funktion der Zeit dar, wie in Abbildung 5.1 gezeigt.
- Beschriften Sie die Grafik.
- Speichern Sie die Grafik als pdf ab.
5.8.4 Zweitstimme bei der Bundestagswahl 2017
Bauen Sie die Grafik nach (Abbildung 5.2).
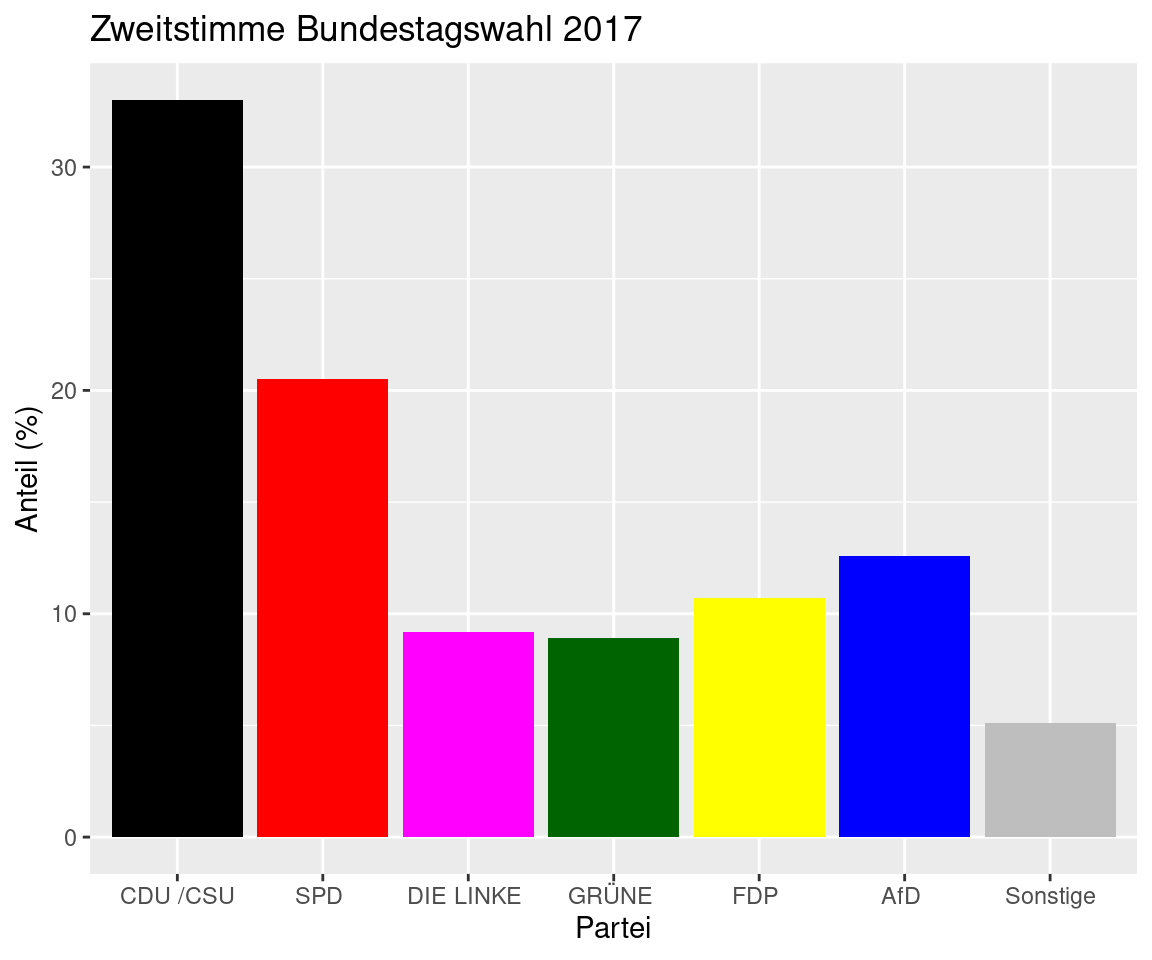
Abbildung 5.2: Zweitstimme bei der Bundestagswahl 2017. Quelle: Der Bundeswahlleiter.
- Lesen Sie den Datensatz Zweitstimme.csv in R ein und ordnen Sie ihn dem Objekt
zweitstimmezu. - Sehen Sie sich den Datensatz an und fassen Sie ihn zusammen.
- Stellen Sie die die Zweitstimmen pro Partei in einem Säulendiagramm dar. Nutzen Sie das geom
geom_col()und lesen Sie den Unterschied zugeom_bar()in der Hilfe nach. Tipps:- Der Variablenname
Zweitstimme 2017enthält ein Leerzeichen. Daher müssen Sie es beim Aufruf zuggplotunbedingt in “`” setzten. - Damit die Parteien in der selben Reihenfolge dargestellt werden, wie im Datensatz angegeben, wandeln Sie die Spalte
Parteiin einfactorum:zweitstimme$Partei <- as_factor(zweitstimme$Partei). - Farben stellen Sie direkt in
geom_col()ein mitfill = c('black', 'red', 'magenta', 'darkgreen', 'yellow', 'blue', 'grey')
- Der Variablenname
- Beschriften Sie die Grafik.
- Speichern Sie die Grafik als pdf ab.
5.8.5 Ergebnisse der Bundestagswahl in einer Grafik
Stellen Sie beide Grafiken untereinander dar wie in Abbildung (5.3) gezeigt.
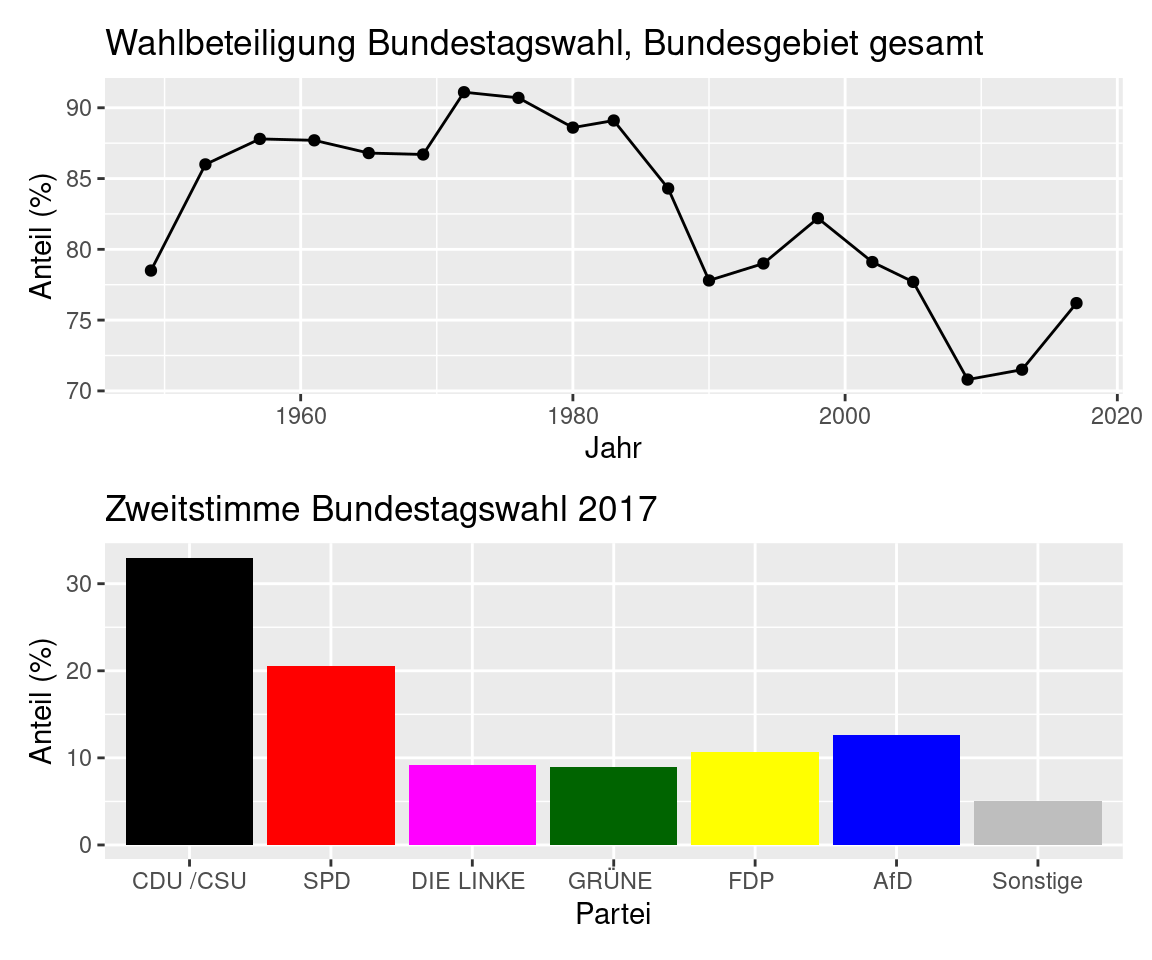
Abbildung 5.3: Ergebnisse der Bundestagswahl 2017. Quelle: Der Bundeswahlleiter.